28 Intervalos de confianza
En este capítulo vamos mostrar como se pueden obtener intervalos de confianza para los parámetros del modelo e intervalos de confianza para predicción.
28.1 Intervalos de confianza para los parámetros
En esta sección se mostrará cómo utilizar la función confint() para obtener intervalos de confianza para los elementos del vector de parámetros \(\boldsymbol{\Theta}\) de un modelo lineal generalizado mixto.
Ejemplo: modelo normal
En este ejemplo su usará la base de datos sleepstudy del paquete lme4 de Bates et al. (2025) sobre el tiempo de reacción promedio por día para un conjunto de individuos en un estudio de privación del sueño. La base de datos contiene la información sobre el tiempo de reacción promedio (Reaction), el número de días de privación del sueño (Days), donde el día 0 corresponde al día en el que los individuos tenían su cantidad normal de sueño, y el número del individuo (en total 18) sobre el que se realizó la observación (Subject). A partir del día 0, hubo una restricción en cada individuo a 3 horas de sueño por noche.

El objetivo es ajustar el siguiente modelo a los datos.
\[\begin{align*} Reaction_{ij} | b_0, b_1 &\sim N(\mu_{ij}, \sigma^2_{Reaction}) \\ \mu_{ij} &= \beta_0 + \beta_1 Days_{ij} + b_{0i} + b_{1i} Days_{ij} \\ \left ( \begin{matrix} b_{0} \\ b_{1} \end{matrix} \right ) &\sim N\left ( \left [ \begin{matrix} 0 \\ 0 \end{matrix} \right ], \left [ \begin{matrix} \sigma^2_{b0} & \sigma_{b01} \\ \sigma_{b01} & \sigma^2_{b1} \end{matrix} \right ] \right ) \end{align*}\]
Lo primero que debemos hacer es ajustar el modelo usando el siguiente código.
## Reaction Days Subject pred_inter_pend_aleatorio modelo_simple
## 1 249.5600 0 308 253.6637 251.4051
## 2 258.7047 1 308 273.3299 261.8724
## 3 250.8006 2 308 292.9962 272.3397
## 4 321.4398 3 308 312.6624 282.8070
## 5 356.8519 4 308 332.3287 293.2742
## 6 414.6901 5 308 351.9950 303.7415
## pred_inter_aleatorio pred_pend_aleatorio
## 1 292.1888 251.4051
## 2 302.6561 271.4918
## 3 313.1234 291.5785
## 4 323.5907 311.6652
## 5 334.0580 331.7519
## 6 344.5252 351.8386La tabla de resultados del modelo ajustado se muestra a continuación.
## Linear mixed model fit by REML ['lmerMod']
## Formula: Reaction ~ Days + (Days | Subject)
## Data: sleepstudy
##
## REML criterion at convergence: 1743.6
##
## Scaled residuals:
## Min 1Q Median 3Q Max
## -3.9536 -0.4634 0.0231 0.4634 5.1793
##
## Random effects:
## Groups Name Variance Std.Dev. Corr
## Subject (Intercept) 612.10 24.741
## Days 35.07 5.922 0.07
## Residual 654.94 25.592
## Number of obs: 180, groups: Subject, 18
##
## Fixed effects:
## Estimate Std. Error t value
## (Intercept) 251.405 6.825 36.838
## Days 10.467 1.546 6.771
##
## Correlation of Fixed Effects:
## (Intr)
## Days -0.138De la tabla anterior podemos ver que las estimaciones del vector de parámetros es
\[ \hat{\boldsymbol{\Theta}} = (\hat{\beta_0}=251.40, \hat{\beta_1}=10.47, \hat{\sigma}_{reaction}=25.59, \hat{\sigma}_{b0}=24.74, \hat{\sigma}_{b1}=5.92, \hat{\rho}_{b0b1}=0.07)^\top \]
Para obtener los intervalos de confianza podemos usar la función confint().
## Computing profile confidence intervals ...## 2.5 % 97.5 %
## .sig01 14.3814379 37.7159918
## .sig02 -0.4815008 0.6849863
## .sig03 3.8011643 8.7533658
## .sigma 22.8982669 28.8579965
## (Intercept) 237.6806955 265.1295145
## Days 7.3586533 13.5759188También es posible obtener los intervalos de confianza por el método boostrap así.
## Computing bootstrap confidence intervals ...##
## 11 message(s): boundary (singular) fit: see help('isSingular')
## 7 warning(s): Model failed to converge with max|grad| = 0.00237033 (tol = 0.002, component 1) (and others)## 2.5 % 97.5 %
## .sig01 12.9637679 35.9428046
## .sig02 -0.5360978 0.8664981
## .sig03 3.1342841 8.4724073
## .sigma 22.6178539 28.5395275
## (Intercept) 237.7970463 265.0299430
## Days 7.3768799 13.7273663Ejemplo: modelo gamma
En este ejemplo analizamos los datos de semiconductores tomados de Myers et al. (2002) sobre un experimento diseñado en una planta de semiconductores. Se emplean seis factores, temperatura de laminación, tiempo de laminación, presión de laminación, temperatura de cocción, tiempo de ciclo de cocción y punto de rocío de cocción, y estamos interesados en la curvatura de los dispositivos de sustrato producidos en la planta. La medición de la curvatura se realiza cuatro veces en cada dispositivo fabricado. Cada variable de diseño se toma en dos niveles. Se sabe que la medida no tiene una distribución normal y las medidas tomadas en el mismo dispositivo están correlacionadas.
Las variables de la base de datos se muestran a continuación.
- Device: Subtrate device
- x1: Lamination Temperature; two levels +1 and -1.
- x2: Lamination Time; two levels: +1 and -1.
- x3: Lamination Presure; two levels: +1 and -1.
- x4: Firing Temperature; two levels: +1 and -1.
- x5: Firing Cycle Time; two levels: +1 and -1.
- x6: Firing Dew Point: two levels: +1 and -1.
- y: Camber measure; in 1e-4 in./in.
![]()
El objetivo es ajustar el siguiente modelo a los datos.
\[\begin{align*} y_{ij} | b_0 &\sim Gamma(\mu_{ij}, \phi) \\ \log(\mu_{ij}) &= \beta_0 + \beta_1 x1_{ij} + \beta_3 x3_{ij} + \beta_5 x5_{ij} + \beta_6 x6_{ij} + b_{0i} \\ b_{0} &\sim N(0, \sigma^2_{b0}) \end{align*}\]
Para ajustar el modelo anterior usamos el siguiente código.
library(hglm)
data(semiconductor)
library(glmmTMB)
fit <- glmmTMB(y ~ x1 + x3 + x5 + x6 + (1 | Device),
data = semiconductor,
family = Gamma(link = log))
summary(fit)## Family: Gamma ( log )
## Formula: y ~ x1 + x3 + x5 + x6 + (1 | Device)
## Data: semiconductor
##
## AIC BIC logLik -2*log(L) df.resid
## -545.2 -530.1 279.6 -559.2 57
##
## Random effects:
##
## Conditional model:
## Groups Name Variance Std.Dev.
## Device (Intercept) 0.02505 0.1583
## Number of obs: 64, groups: Device, 16
##
## Dispersion estimate for Gamma family (sigma^2): 0.101
##
## Conditional model:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) -4.69447 0.05635 -83.31 < 2e-16 ***
## x1 0.18290 0.05629 3.25 0.001158 **
## x3 0.30496 0.05629 5.42 6.04e-08 ***
## x5 -0.19565 0.05632 -3.47 0.000513 ***
## x6 -0.36961 0.05630 -6.57 5.19e-11 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1De la tabla anterior podemos ver que las estimaciones del vector de parámetros es
\[ \hat{\boldsymbol{\Theta}} = (\hat{\beta_0}=-4.71, \hat{\beta_1}=0.18, \hat{\beta_3}=0.31, \hat{\beta_5}=-0.19, \hat{\beta_6}=-0.37, \hat{\phi}=0.31, \hat{\sigma}_{b0}=0.17)^\top \]
Para obtener los intervalos de confianza usamos las siguientes instrucciones.
FUN <- function(model=fit) {
temp_mod <- refit(fit, simulate(fit)[[1]])
result <- c(as.numeric(temp_mod$fit$par[1:7]),
temp_mod$obj$report()$sd[[1]])
return(result)
}
b1 <- lme4::bootMer(fit, FUN, nsim=100, .progress="txt")Para imprimir los intervalos de confianza de los 8 parámetros hacemos lo siguiente:
28.2 Intervalos de confianza para predicción
En esta sección se mostrará cómo utilizar el paquete ggeffects de Lüdecke (2025) para obtener intervalos de confianza para predicciones de un modelo lineal generalizado mixto.
Ejemplo: modelo normal
En este ejemplo vamos a retomar los datos sleepstudy del paquete lme4 de Bates et al. (2025) sobre el tiempo de reacción promedio por día para un conjunto de individuos en un estudio de privación del sueño. La base de datos contiene la información sobre el tiempo de reacción promedio (Reaction), el número de días de privación del sueño (Days), donde el día 0 corresponde al día en el que los individuos tenían su cantidad normal de sueño, y el número del individuo (en total 18) sobre el que se realizó la observación (Subject). A partir del día 0, hubo una restricción en cada individuo a 3 horas de sueño por noche.

Vamos a mostrar gráficamente los datos.
library(ggplot2)
ggplot(data = sleepstudy, aes(x = Days, y = Reaction, color = Subject)) +
geom_point() +
theme_bw() +
facet_wrap(~ Subject) + labs(y = "Reaction time") +
theme(legend.position = "none")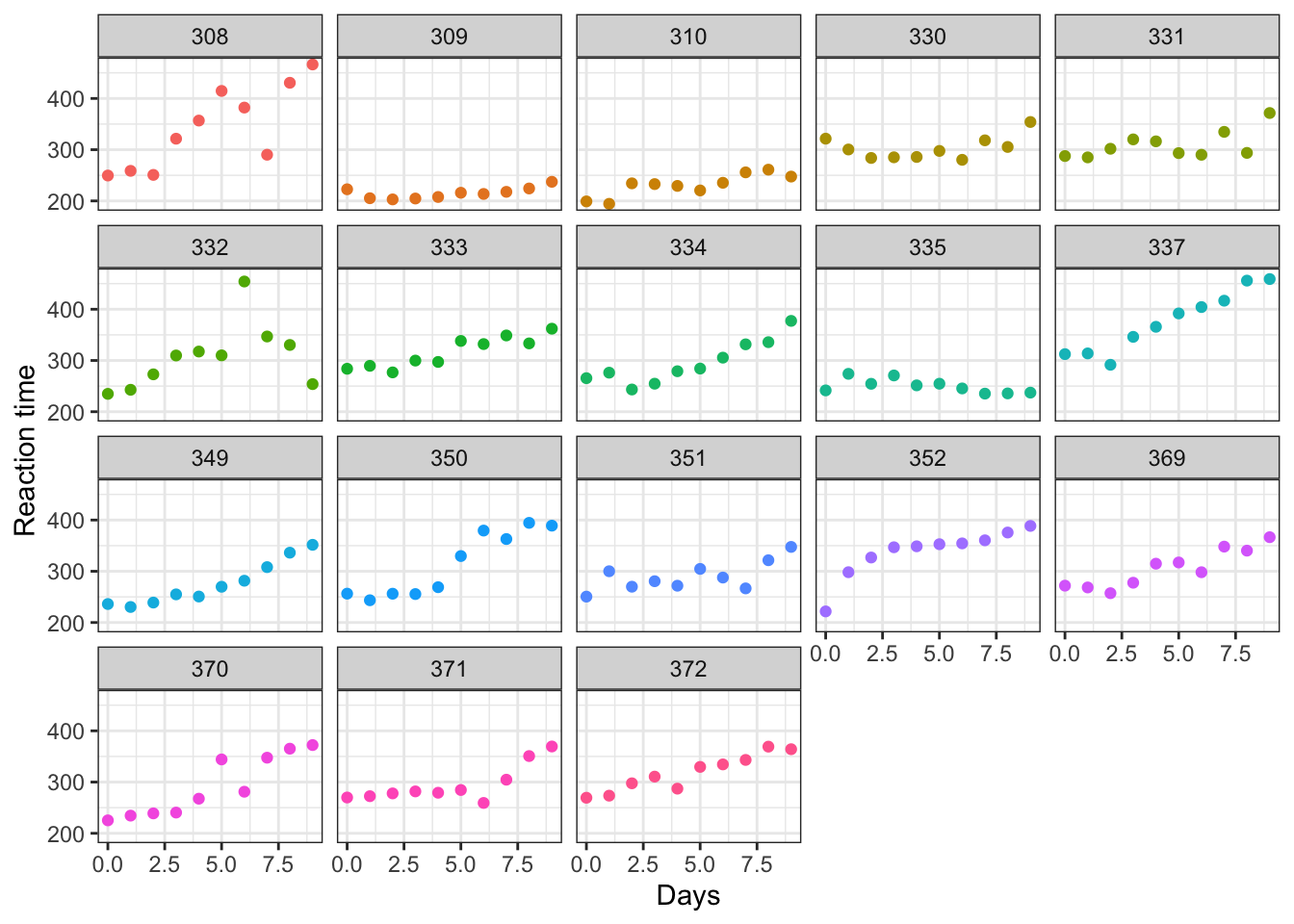
Ahora vamos a ajustar el modelo de interés.
library(ggeffects)
library(lme4)
data(sleepstudy)
fit <- lmer(Reaction ~ Days + (1 + Days | Subject), data = sleepstudy)Predicción para cada individuo
Ahora supongamos que nos interesa predecir el tiempo de reacción de los pacientes 335 y 337 en los días de observación 1, 5 y 9, y en los días 12, 15 que no fueron aún observados. Adicionalmente queremos los intervalos de confianza del 95% para la predicción en esos mismos días. Para eso vamos a usar la función predict_response() de la siguiente manera:
pre1 <- predict_response(model=fit,
ci_level=0.95,
terms=c("Days [1, 5, 9, 12, 15]",
"Subject [335, 337]"),
type="random")
pre1## # Predicted values of Reaction
##
## Subject: 335
##
## Days | Predicted | 95% CI
## ---------------------------------
## 1 | 250.79 | 237.39, 264.18
## 5 | 249.65 | 230.74, 268.56
## 9 | 248.51 | 219.63, 277.38
## 12 | 247.65 | 210.42, 284.88
## 15 | 246.80 | 200.91, 292.69
##
## Subject: 337
##
## Days | Predicted | 95% CI
## ---------------------------------
## 1 | 305.39 | 292.00, 318.79
## 5 | 381.77 | 362.86, 400.68
## 9 | 458.16 | 429.28, 487.03
## 12 | 515.44 | 478.21, 552.67
## 15 | 572.73 | 526.84, 618.62Como resultado se obtienen dos tablas, una para el paciente 335 y otra para el paciente 337. Cada tabla muestra los días, la predicción del tiempo de reacción promedio \(\mu\) y los intervalos de confianza del 95%.
Es posible representar gráficamente la predicción y los intervalos usando la función genérica plot() sobre el objeto pre1 así:
## Data points may overlap. Use the `jitter` argument to add some amount of
## random variation to the location of data points and avoid overplotting.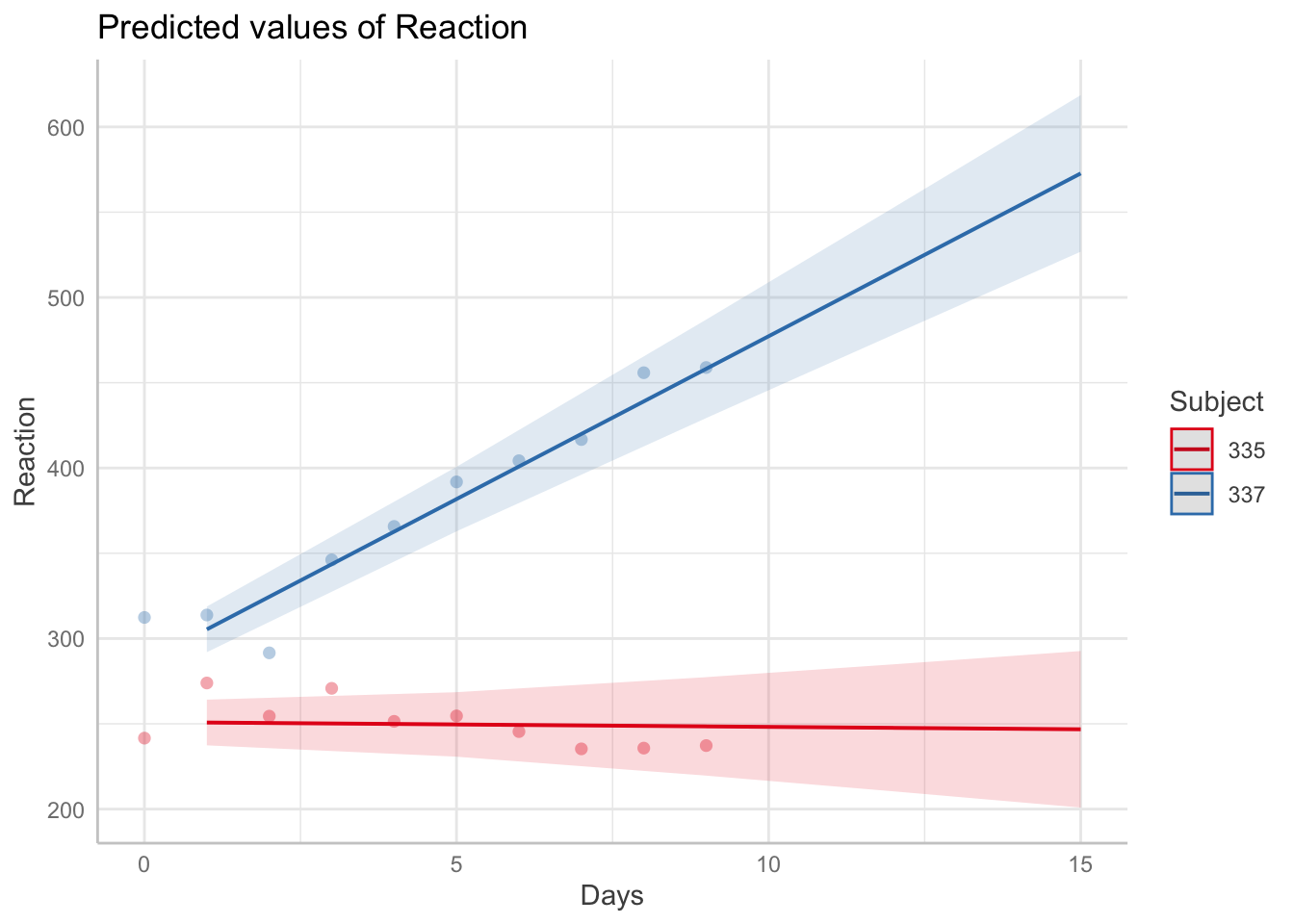
En la figura anterior se observan los modelos ajustados representados por las líneas continuas y los intervalos como sombras. Adicionalmente se observan unos puntos que corresponden a los datos originales para los pacientes considerados.
La figura anterior puede ser usada para monitorear la variable respuesta en los días 10, 11, 12 y siguientes. Los intervalos de confianza se hacen más anchos a medida que el número de días aumenta, esto es esperado ya que si queremos hacer una estimación lejos del día 9, muy seguramente tendremos más incertidumbre que para días cercanos al 9.
Predicciones condicionales
Las predicciones condicionales son predicciones para el parámetro \(\mu\) de la variable respuesta pensando en un paciente (o grupo) “típico”, es decir, predicciones para \(\mu\) sin incluir los efectos aleatorios predichos en la ecuación. Para obtener esta predicción se usa la función predict_response con margin="mean_reference".
Supongamos que nos interesa obtener predicciones condicionales con respecto a la variable días fijada en los valores de 1, 5, 9 y 12. El código para realizar esto es el siguiente:
# conditional predictions
pre2 <- predict_response(model=fit,
terms="Days [1, 5, 9, 12]",
margin="mean_reference")
pre2## # Predicted values of Reaction
##
## Days | Predicted | 95% CI
## ---------------------------------
## 1 | 261.87 | 248.48, 275.27
## 5 | 303.74 | 284.83, 322.65
## 9 | 345.61 | 316.74, 374.48
## 12 | 377.01 | 339.78, 414.24
##
## Adjusted for:
## * Subject = 0 (population-level)Como resultado obtenemos una tabla con el valor predicho para \(\mu\) en los días solicitados y los intervalos de confianza.
Es posible representar estos resultados gráficamente de la siguiente manera.
## Data points may overlap. Use the `jitter` argument to add some amount of
## random variation to the location of data points and avoid overplotting.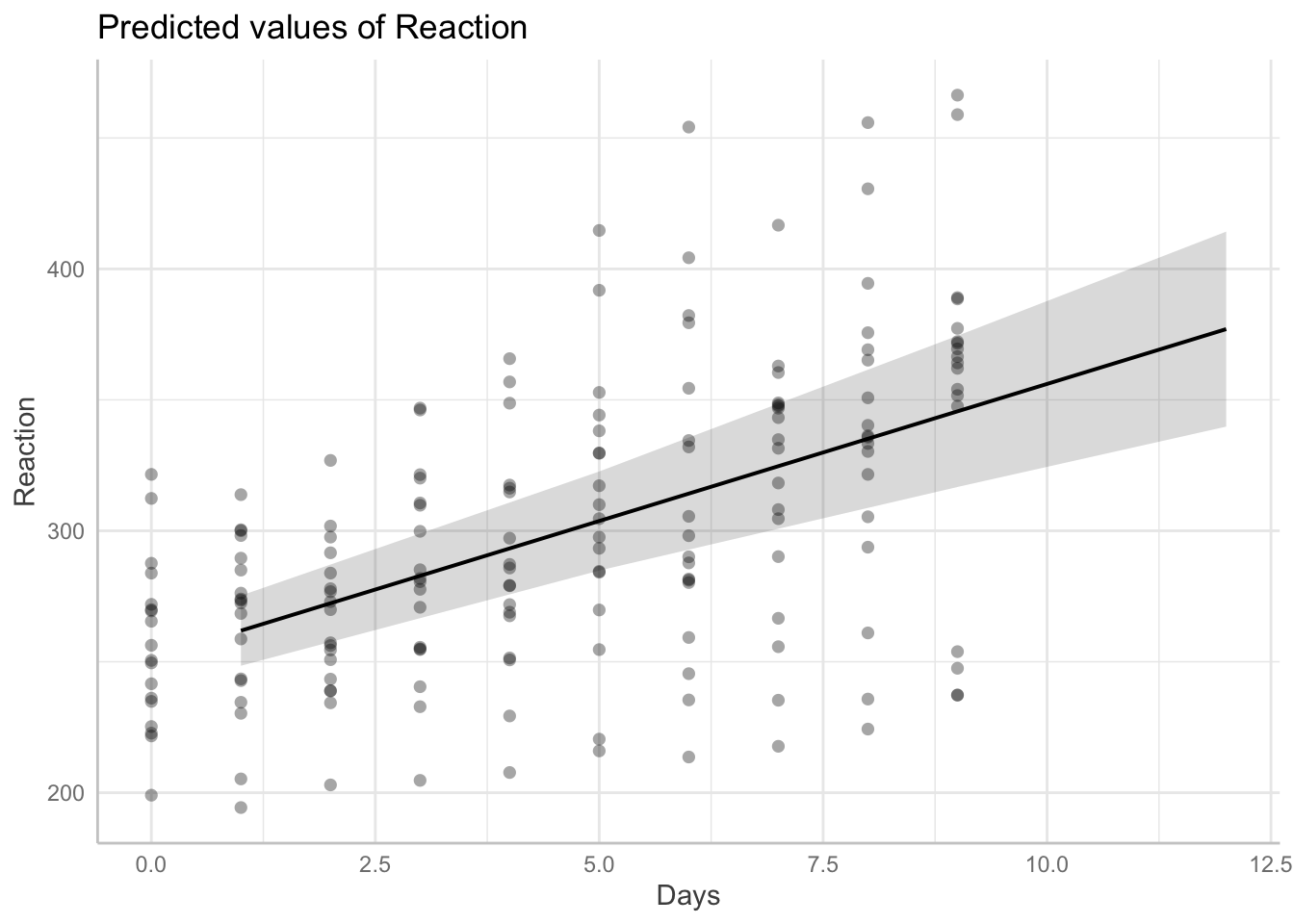
Predicciones marginales
Las predicciones marginales son predicciones para el parámetro \(\mu\) de la variable respuesta pensando en el efecto de una covariable \(X\) sobre todos los pacientes (o grupos) de forma general.
# average marginal predictions
pre3 <- predict_response(model=fit,
terms="Days [1, 5, 9]",
margin="empirical")
pre3## # Average predicted values of Reaction
##
## Days | Predicted | 95% CI
## ---------------------------------
## 1 | 261.87 | 248.48, 275.27
## 5 | 303.74 | 284.83, 322.65
## 9 | 345.61 | 316.74, 374.48Como resultado obtenemos una tabla con el valor predicho para \(\mu\) en los días solicitados y los intervalos de confianza.
Es posible representar estos resultados gráficamente de la siguiente manera.
## Data points may overlap. Use the `jitter` argument to add some amount of
## random variation to the location of data points and avoid overplotting.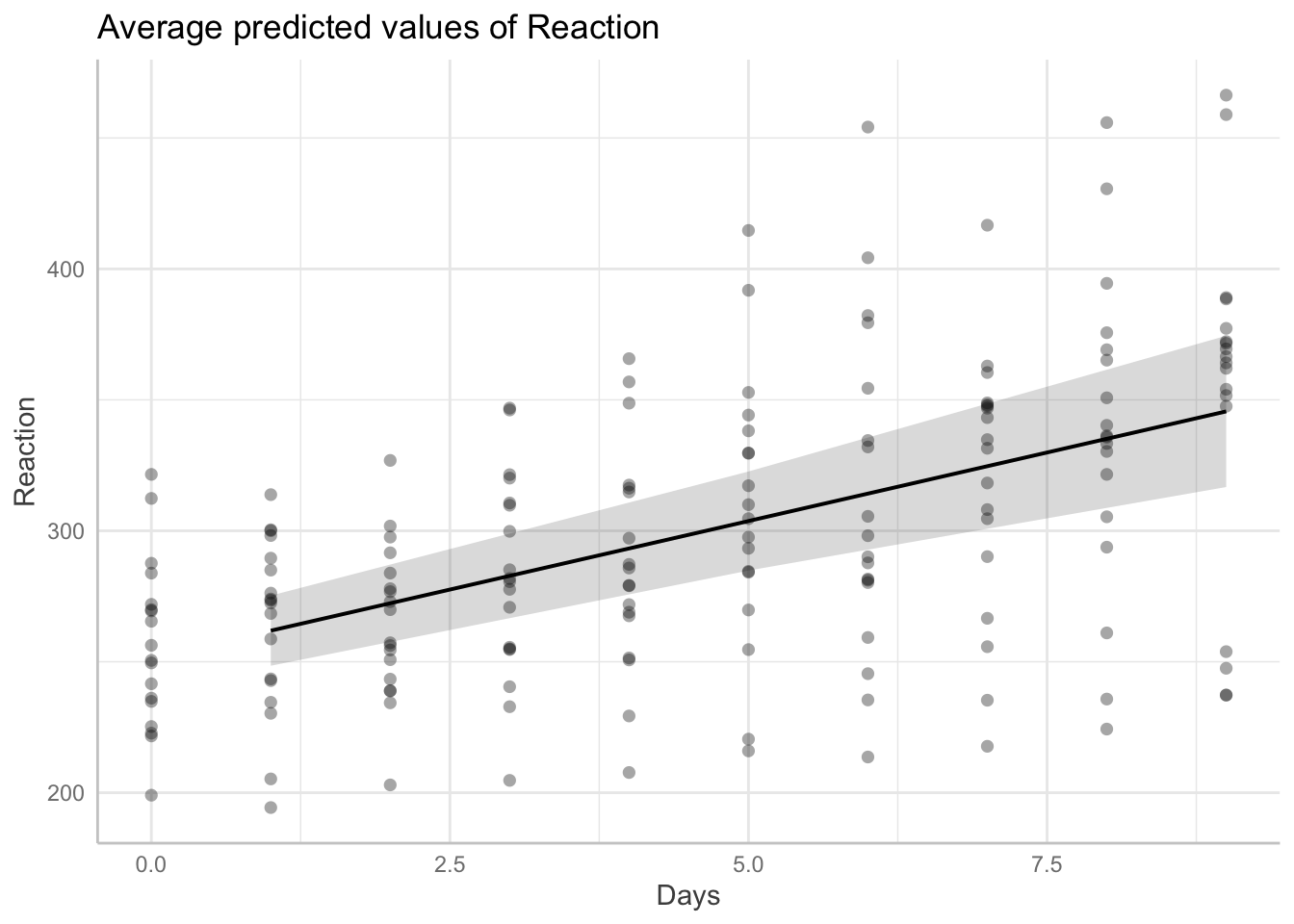
Ver https://www.andrewheiss.com/blog/2022/11/29/conditional-marginal-marginaleffects/